Los pequeños compuestos moleculares desempeñan un papel crucial en la salud humana, desde hormonas de crecimiento hasta fármacos oncológicos. La monitorización de estas moléculas es esencial para garantizar un correcto control de las dosis y para que los pacientes puedan supervisar sus condiciones médicas en casa. Sin embargo, la detección de estas pequeñas moléculas se enfrenta a desafíos significativos.
A pesar de que se han desarrollado sensores para detectar algunas de estas moléculas, la mayoría de ellos están limitados a entornos de investigación y descubrimiento de fármacos, y solo pueden identificar un rango restringido de moléculas con características específicas. Existe, por tanto, una necesidad apremiante de sensores capaces de detectar y señalar la presencia de diversas pequeñas moléculas con diferentes formas, tamaños, flexibilidad y polaridad.
Innovación a través de la inteligencia artificial
Un equipo de científicos liderado por el ganador del Premio Nobel David Baker, de la Universidad de Washington, ha desarrollado un método computacional que permite diseñar proteínas capaces de unirse y señalar una amplia gama de pequeñas moléculas con gran eficacia. Baker recibió el Nobel de Química en 2024 por sus innovaciones en el diseño computacional de proteínas.
La investigación, publicada en la revista Science, se realizó en parte en el Advanced Photon Source (APS), una instalación del Departamento de Energía de EE. UU. en el Laboratorio Nacional de Argonne. Este enfoque demuestra cómo las estrategias de diseño modular pueden resolver el desafío de crear sensores de proteínas para pequeñas moléculas, que deben unirse a estas moléculas y luego emitir una señal de su presencia.
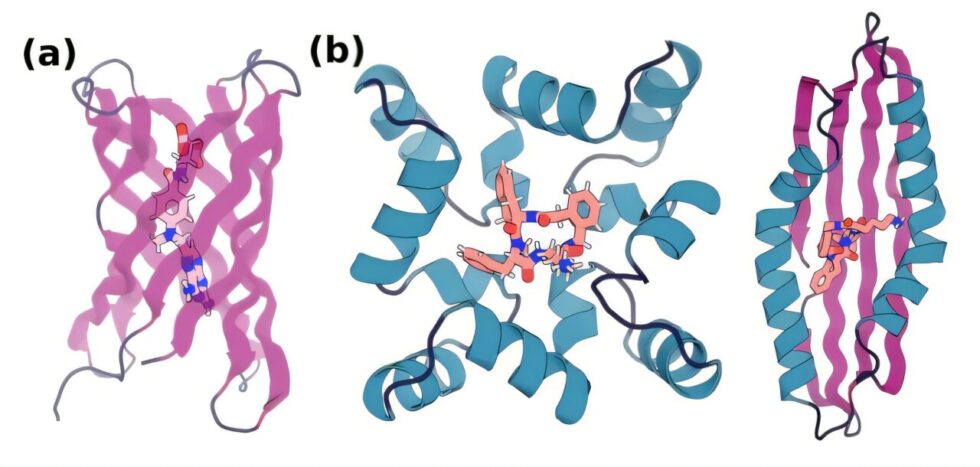
Los científicos emplearon un algoritmo de aprendizaje automático basado en AlphaFold2, un predictor de estructuras proteicas, para generar miles de proteínas diseñadas para unirse a diferentes ligandos, como el ácido cólico, un biomarcador de enfermedades hepáticas, y el metotrexato, un fármaco oncológico que requiere un seguimiento regular. Tras el diseño computacional, se probaron las proteínas en el laboratorio, identificando uniones específicas a ligandos mediante métodos de aprendizaje automático.
Para validar la precisión de su enfoque, el equipo utilizó la cristalografía de rayos X en el APS, lo que les permitió confirmar que las estructuras predichas coincidían con la realidad. Este proceso de verificación es fundamental, dado que los algoritmos de predicción, aunque excelentes, necesitan ser respaldados por datos estructurales concretos.
El siguiente paso fue transformar las proteínas de unión en proteínas de señalización. Gracias a su modularidad, los científicos pudieron crear dos tipos diferentes de eventos de señalización, que permiten aplicaciones en salud, como la regulación de la liberación de fármacos en terapias contra el cáncer. También se desarrollaron proteínas que pueden detectar microplásticos y toxinas medioambientales, ampliando así el potencial uso de esta tecnología más allá de la biomedicina.
Con el desarrollo de un mayor número de uniones de alta afinidad y especificidad, se podrían facilitar pruebas rápidas en casa para detectar enfermedades hepáticas, ciertos tipos de cáncer y condiciones tiroideas. Esta innovación no solo representa un avance en el campo de la salud, sino que también pone de manifiesto la capacidad de la ciencia para abordar problemas complejos de manera creativa y efectiva.







