Descubriendo los mecanismos de arresto en la síntesis de proteínas
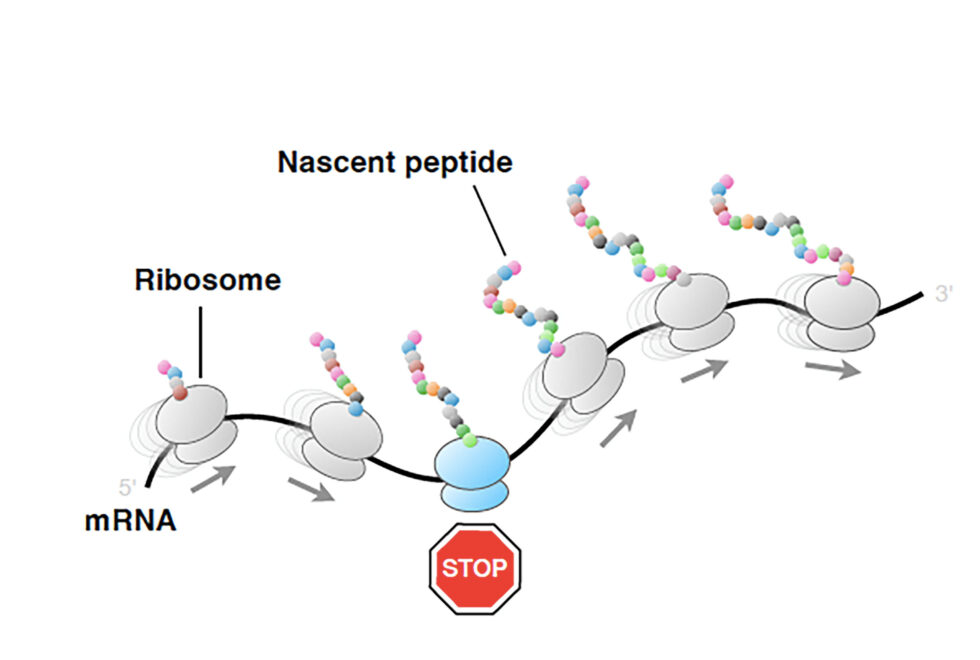
La síntesis de proteínas es un proceso fundamental en la biología celular, donde los ribosomas actúan como fábricas que traducen la información genética en polipéptidos funcionales. Sin embargo, este proceso puede verse obstaculizado por elementos conocidos como péptidos de arresto del ribosoma (RAPs, por sus siglas en inglés), cuya comprensión ha sido un desafío en el ámbito de la biología molecular. Recientemente, un equipo de investigadores japoneses ha logrado identificar y caracterizar estructuralmente estos RAPs en Escherichia coli, aportando nuevos conocimientos sobre los mecanismos que regulan la síntesis de proteínas.
El ribosoma, cuya función va más allá de la mera síntesis de proteínas, también desempeña un papel crucial en la regulación del proceso de traducción. Este se inicia cuando el ribosoma reconoce el sitio de inicio y comienza a ensamblar los aminoácidos en la secuencia polipeptídica correspondiente. Sin embargo, en determinados casos, ciertos péptidos nascentes pueden interactuar con el túnel ribosómico, alterando su estructura interna y provocando el estancamiento de la elongación, fenómeno conocido como «arresto de traducción». En este contexto, los RAPs, que son codificados por marcos de lectura abiertos pequeños (sORFs), emergen como factores importantes en la regulación de la expresión génica.
El Dr. Yuhei Chadani, profesor asociado en la Facultad de Ciencias Ambientales, Vida, Naturales y Tecnología de la Universidad de Okayama, junto con su equipo, se propuso desentrañar estos mecanismos mediante el análisis de 38 sORFs en E. coli. De estos, 18 sORFs mostraron ser capaces de inducir inhibición del crecimiento celular, aunque sus efectos citotóxicos no estaban relacionados con la regulación de genes descendentes. Este hallazgo resalta la complejidad de los mecanismos reguladores que operan a nivel molecular, y la necesidad de profundizar en su estudio.
La investigación también reveló que la sobreexpresión de ciertos RAPs, como el TnaC, puede inducir la expresión de proteínas de choque por frío (CSPs), que se activan como respuesta a la inhibición de la elongación de la traducción. A través de un análisis proteómico comparativo, los investigadores encontraron que tanto el arresto de traducción mediado por TnaC como el inducido por antibióticos estaban asociados con la expresión de CSPs, lo que sugiere un vínculo entre el estrés ambiental y la regulación de la síntesis de proteínas.
Un aspecto destacado del estudio es la identificación de los péptidos arrestadores PepNL y NanCL, que también indujeron el arresto de traducción en E. coli. A través del análisis estructural, se determinó que el péptido PepNL adopta una conformación estable en forma de mini-horquilla dentro del túnel ribosómico, lo que impide la terminación normal de la traducción. Este descubrimiento es significativo, ya que implica que el PepNL no requiere un inductor de arresto para su actividad, a diferencia de otros RAPs conocidos.
Estos hallazgos no solo enriquecen nuestra comprensión de los mecanismos de regulación de la síntesis de proteínas, sino que también abren nuevas vías para el desarrollo de biosensores basados en péptidos reguladores. Como concluye el Dr. Chadani, «nuestras aproximaciones para identificar PepNL y NanCL, así como el mecanismo molecular distinto de estancamiento y regulación de la traducción, proporcionan valiosos insights para descifrar los códigos genéticos ocultos en las secuencias polipeptídicas». Esta investigación, publicada en la revista Nature Communications, subraya la relevancia de la biología molecular en la búsqueda de soluciones innovadoras a desafíos biotecnológicos contemporáneos.