Un nuevo estudio publicado en la revista Biochemistry aporta información valiosa sobre cómo las bacterias regulan sus genes, desafiando ideas preconcebidas sobre el comportamiento de las proteínas. La investigación se centra en la comparación del uso de una molécula señalizadora llamada AMP cíclico (cAMP) por parte de dos especies bacterianas: Escherichia coli y Mycobacterium tuberculosis.
Regulación alostérica en proteínas receptoras de cAMP
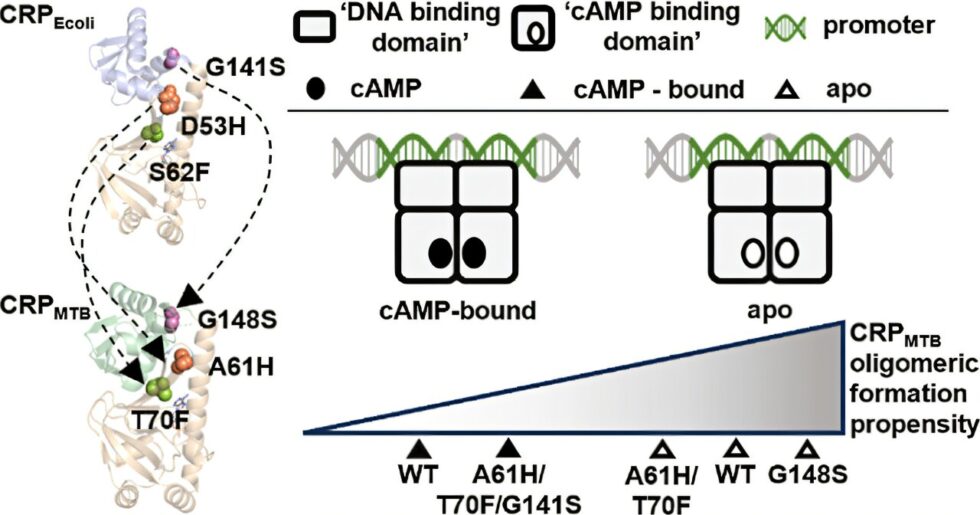
El equipo de investigación ha proporcionado claves sobre cómo las proteínas receptoras de cAMP (CRP) en bacterias responden de forma diferente a esta molécula señalizadora. Al comparar la regulación alostérica de la CRP de Escherichia coli (CRPEcoli) y la CRP de Mycobacterium tuberculosis (CRPMTB), los investigadores cuestionan la suposición de que la similitud estructural predice el comportamiento funcional en proteínas alostéricas.
El estudio introdujo mutaciones homólogas estructuralmente de hotspots alostéricos previamente identificados en CRPEcoli en CRPMTB para investigar sus efectos en la estructura, estabilidad y función de la proteína. Los resultados revelan que, a pesar de su semejanza estructural, estas dos proteínas de bacterias distantes no comparten comportamientos alostéricos idénticos ni puntos calientes en respuesta a señales moleculares. Estos hallazgos subrayan la complejidad de la regulación alostérica y sugieren que los mecanismos específicos de cada especie son los que impulsan la regulación genética en bacterias.
Más allá de avanzar en el conocimiento fundamental sobre la señalización bacteriana, esta investigación tiene implicaciones potenciales para el desarrollo de fármacos. Comprender las propiedades alostéricas únicas de CRPMTB puede informar estrategias terapéuticas que apunten a reguladores transcripcionales en M. tuberculosis, un factor crítico en la adaptación y patogénesis bacteriana.
El estudio, titulado «Identificación de Hotspots Alostéricos en la Proteína Receptora de cAMP de Mycobacterium tuberculosis a través de la Homología Estructural», fue liderado por el Dr. Rodrigo Maillard en la Universidad de Georgetown. La investigación futura explorará cómo las diferencias evolutivas moldean las vías de señalización alostérica entre las especies bacterianas. Comprender los mecanismos regulatorios únicos de CRP en M. tuberculosis en comparación con E. coli podría ofrecer información valiosa sobre la adaptación bacteriana y la regulación genética.
Adicionalmente, la identificación de nuevos elementos regulatorios dentro de las vías mediadas por CRP podría informar estrategias antimicrobianas destinadas a interrumpir selectivamente redes de señalización bacteriana esenciales, lo que podría llevar a enfoques terapéuticos más específicos.
Más información:
Stephen P. Dokas et al, Identifying Allosteric Hotspots in Mycobacterium tuberculosis cAMP Receptor Protein through Structural Homology, Biochemistry (2025). DOI: 10.1021/acs.biochem.4c00723
Proporcionado por
Georgetown University Medical Center