Recientemente, el ribosoma, una de las máquinas moleculares más antiguas desde el punto de vista evolutivo, ha sido reconocido como un regulador activo de la expresión génica a nivel de biosíntesis de proteínas. Este proceso es fundamental para el desarrollo y la función celular, ya que implica la conversión de la información genética en proteínas. La etapa final de este proceso, en la que la información codificada en el ARN mensajero (ARNm) se transfiere, se conoce como traducción.
El grupo de investigación «Inmunobiología y Bioquímica» liderado por la profesora Kathrin Leppek en el Instituto de Química Clínica y Farmacología Clínica (IKCKP) en el UKB está investigando el control de la traducción a través de la interacción directa del ribosoma con los ARNm. Según la profesora Leppek, miembro del Cluster de Excelencia InmunoSensation2 en la Universidad de Bonn, «como maquinaria central de la traducción esencial para toda forma de vida, el ribosoma y los factores asociados, como proteínas o estructuras de ARN, son el foco de nuestro interés de investigación». Hay evidencia creciente de que la composición del ribosoma influye en la traducción selectiva, de tal manera que ribosomas personalizados se unen y traducen preferentemente ciertos ARNm.
El papel de los IRES en la expresión génica
Entre las estructuras investigadas, los investigadores de Bonn han analizado los sitios de entrada ribosómica internos (IRES, por sus siglas en inglés). Estas son secuencias especializadas que se pliegan dentro de una cadena de ARN y son particularmente conocidas en el material genético de los virus, que utilizan estas estructuras para secuestrar los ribosomas del huésped tras una infección. Por ejemplo, el virus de la hepatitis C o el poliovirus pueden iniciar la producción de nuevas proteínas virales independientemente de todos los factores de iniciación gracias a sus elementos IRES.
Los elementos IRES permiten la iniciación de la traducción de manera independiente del capuchón 5′ del ARNm, que es un capuchón protector que equipan las propias cadenas de ARNm del huésped. Este capuchón permite la traducción en condiciones normales, pero se bloquea durante una infección viral.
Los IRES fueron descritos por primera vez en el genoma viral, donde permiten la replicación de virus en células infectadas al reclutar ribosomas del huésped. Sin embargo, en las últimas décadas se han descrito cada vez más IRES en células eucariotas, que, a diferencia de los virus, poseen un núcleo. «Esto refuerza la visión general de que estos elementos también están involucrados en la regulación de la traducción en células eucariotas», afirma Philipp Koch, miembro del grupo de investigación de Leppek y autor principal del artículo publicado en The EMBO Journal.
El colega y coautor Martin Haimann, también estudiante de doctorado en la Universidad de Bonn, añade: «Sin embargo, un desafío importante ha sido la caracterización exacta y confiable de los IRES recientemente descritos, especialmente de los ARNm eucariotas, que ha resultado difícil debido a obstáculos técnicos y artefactos asociados con las tecnologías existentes».
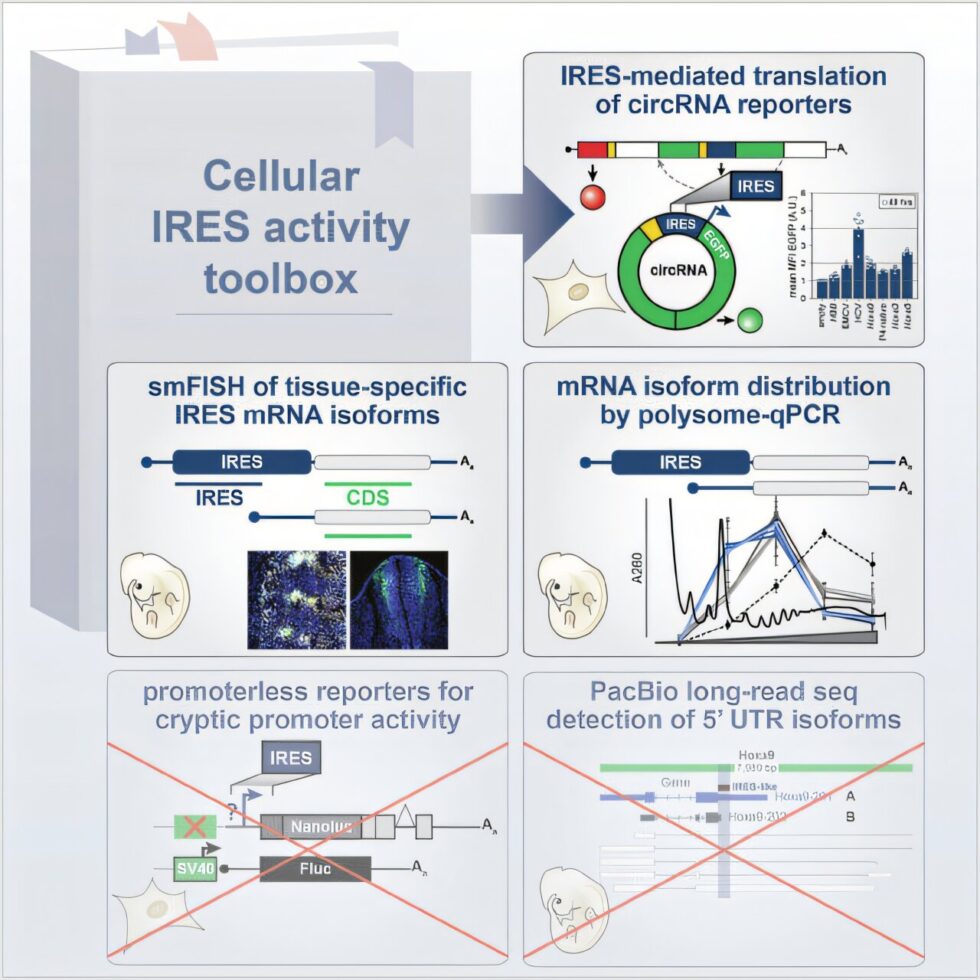
En su trabajo de investigación actual, los investigadores de Bonn han recopilado y probado una serie de técnicas versátiles que permitirán, en el futuro, la caracterización robusta de los IRES. Uno de los métodos importantes implica el uso de reporteros de ARN circular, que pueden confirmar la actividad mediada por IRES de elementos de ARN. Otras técnicas incluyen técnicas de tinción cuantitativa de ARNm individuales en tejido embrionario de ratón y la determinación de la tasa de traducción de ARNm que contienen IRES.
Según Koch, «un conjunto de herramientas tan completo que se puede aplicar en células cultivadas y tejido embrionario representa un nuevo estándar de oro para la prueba y caracterización robusta de los IRES». La profesora Leppek añade que «los elementos IRES fuertes son directamente relevantes para la biología sintética y las terapias emergentes basadas en ARNm».