Los proteínas son los bloques fundamentales de la vida, compuestas por cadenas de péptidos que a su vez se forman a partir de una secuencia de aminoácidos. Desempeñan funciones cruciales, desde la estabilización de la estructura celular hasta la catálisis de reacciones químicas. La diversidad de proteínas se incrementa mediante modificaciones que ocurren después de la síntesis de las cadenas peptídicas.
Una de estas modificaciones es el empalme proteico. En este proceso, una parte de la proteína, conocida como «inteína», se elimina de la cadena peptídica para garantizar el correcto plegamiento y funcionamiento de la proteína final. Investigadores de la Universidad de Münster, liderados por el químico de proteínas Prof. Henning Mootz y el estudiante de doctorado Christoph Humberg, han abordado una pregunta de larga data en este ámbito: ¿por qué una variante especial de las inteínas, las «split inteins», frecuentemente presenta problemas en el laboratorio que reducen significativamente la eficiencia de la reacción? Su investigación ha identificado el mal plegamiento de las proteínas como una de las causas y han desarrollado un método para prevenirlo.
El estudio, publicado en la revista Nature Communications, destaca la relevancia del empalme proteico en la investigación, a pesar de que raramente se produce de forma natural. La solución encontrada por el equipo de Münster abre nuevas posibilidades para utilizar las split inteins en la producción de proteínas útiles tanto en la investigación básica como en aplicaciones en biotecnología y biomedicina.
Avances en la producción de proteínas complejas
Científicos de todo el mundo están trabajando intensamente en la síntesis de proteínas complejas a partir de dos fragmentos que son difíciles o imposibles de producir mediante métodos convencionales. De este modo, se pueden obtener proteínas quiméricas en las que, por ejemplo, una parte se ha producido en células de mamíferos, mientras que la otra ha sido sintetizada químicamente, modificada selectivamente o obtenida de células bacterianas.
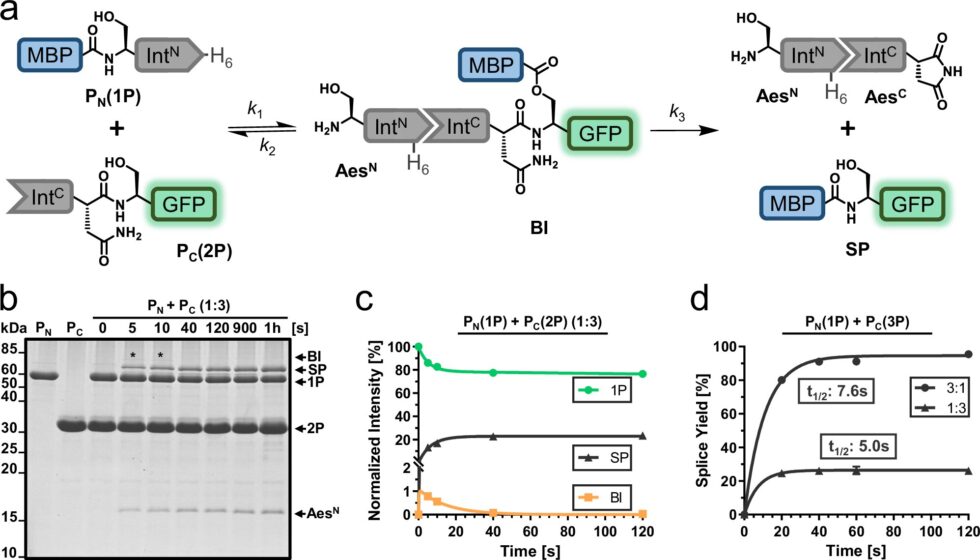
Para llevar a cabo esta tarea, se requieren split inteins particularmente potentes como herramienta. Estas inteínas pueden unir partes separadas de proteínas, ya que están compuestas por dos fragmentos que se localizan en las cadenas peptídicas inicialmente separadas. Una vez que las partes se unen, la split intein se elimina automáticamente.
En su investigación, el equipo examinó la «Aes intein», que permite una amplia gama de aplicaciones gracias a una forma rara de catálisis. A pesar de que ambos fragmentos de la split intein se produjeron en laboratorio en células bacterianas, mostraron una baja productividad, similar a la de otras inteínas. Mediante métodos cromatográficos y biofísicos, el equipo descubrió que una gran proporción de uno de los fragmentos producidos se encontraba en forma de un agregado proteico inactivo con un plegamiento específico incorrecto. A partir de estos hallazgos, los investigadores llegaron a conclusiones sobre la causa del mal plegamiento y utilizaron análisis bioinformáticos para identificar algunos aminoácidos responsables del mismo.
Con el uso de métodos biológicos moleculares, introdujeron mutaciones seleccionadas en el fragmento de la inteína, lo que casi suprimió completamente la formación de agregados y aumentó la productividad de la split intein de manera notable. Este avance representa un paso significativo en la mejora de métodos para la producción de proteínas de diseño, lo que podría tener un impacto profundo en diversas áreas de la ciencia y la medicina.
Más información: Christoph Humberg et al, A cysteine-less and ultra-fast split intein rationally engineered from being aggregation-prone to highly efficient in protein trans-splicing, Nature Communications (2025). DOI: 10.1038/s41467-025-57596-x