Un equipo de ingenieros de la Universidad de California en San Diego ha desarrollado una herramienta innovadora llamada ROADIES, que promete revolucionar la manera en que los investigadores inferen las relaciones evolutivas entre especies. Este avance es especialmente relevante en un contexto donde la biología y la medicina dependen cada vez más de la comprensión de las interconexiones entre diversas especies.
ROADIES, que significa «Estimación de Árboles de Especies Libre de Referencia, Libre de Ortología y Consciente de Discordancia», se presenta como una solución escalable, automatizada y accesible para los científicos. Permite inferir árboles de especies directamente a partir de datos genómicos en bruto, reduciendo la necesidad de conocimientos específicos y recursos computacionales que tradicionalmente han sido un obstáculo para muchos investigadores.
Innovaciones en la inferencia de especies
La importancia de los árboles de especies radica en su capacidad para solidificar nuestro entendimiento sobre la evolución de las especies, lo que a su vez puede facilitar la identificación de regiones funcionales del genoma que podrían servir como dianas para fármacos, vincular características físicas a cambios genómicos y guiar esfuerzos de conservación.
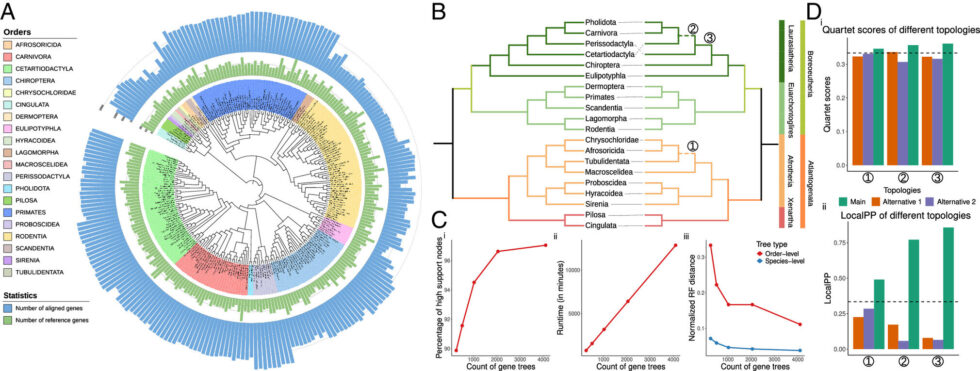
En un estudio publicado en la revista Proceedings of the National Academy of Sciences, el equipo liderado por el profesor Yatish Turakhia demostró que ROADIES genera árboles de especies de calidad comparable con los métodos de vanguardia actuales, pero en una fracción del tiempo y esfuerzo requeridos. Aunque el estudio se centró en cuatro formas de vida diversas—mamíferos placentarios, moscas de la fruta, aves y levaduras—ROADIES es aplicable a cualquier especie.
Los investigadores destacan que la rápida evolución de las técnicas de secuenciación de alto rendimiento ha permitido la producción masiva de ensamblajes genómicos. Sin embargo, la inferencia precisa de árboles de especies ha permanecido fuera del alcance de muchos debido a su complejidad. ROADIES se posiciona como una solución transformadora que promete simplificar este proceso, haciéndolo accesible a un mayor número de científicos y aplicaciones.
Una de las innovaciones clave de ROADIES es su capacidad para operar sin depender de regiones genómicas predefinidas, como los genes codificadores de proteínas. En lugar de ello, la herramienta utiliza una selección aleatoria de loci de los genomas de entrada, lo que elimina la necesidad de anotación genómica previa, un paso que suele ser engorroso y que limita la eficiencia de otros métodos.
Además, ROADIES se beneficia de la existencia de genes que aparecen en múltiples copias en el genoma, un fenómeno común en muchas especies. Al integrar métodos desarrollados en la misma universidad, ROADIES evita la necesidad de inferir ortología, es decir, determinar la correspondencia de copias de genes individuales en diferentes especies, lo que representa un avance significativo en la automatización del proceso.
Este enfoque no solo elimina pasos complicados, sino que también reduce considerablemente la potencia computacional requerida en comparación con herramientas existentes. La investigación enfatiza la escalabilidad de ROADIES, que puede manejar conjuntos de datos con cientos de genomas, produciendo filogenias que son concordantes con estudios a gran escala realizados por expertos, pero con una fracción del tiempo y esfuerzo invertidos.
Los investigadores continúan trabajando en mejorar las capacidades de ROADIES, incluyendo la incorporación de nuevos taxones en árboles de especies existentes y la posible utilización de unidades de procesamiento gráfico (GPUs) para permitir el procesamiento de decenas de miles de genomas. De cara al futuro, la ambición es que ROADIES esté preparado para afrontar los grandes proyectos de secuenciación que se están llevando a cabo para secuenciar miles de especies, y eventualmente, cada especie eucariótica existente en la Tierra.