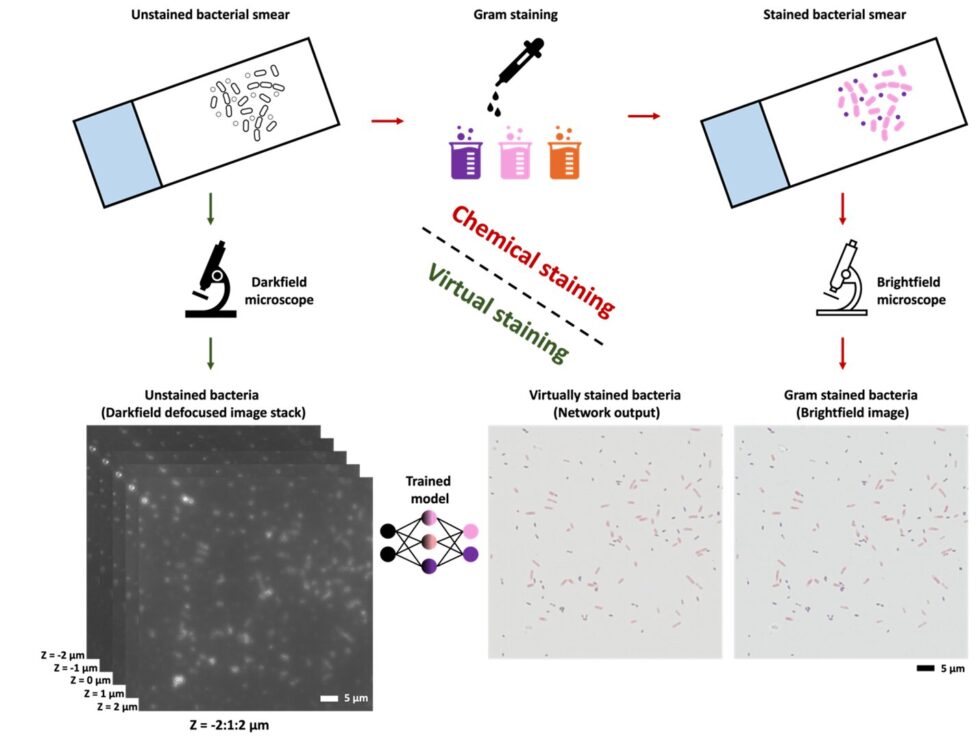
Un equipo de investigadores de la Universidad de California, Los Ángeles (UCLA) ha desarrollado una técnica de tinción virtual de Gram que promete revolucionar la forma en que los microbiológos tiñen y clasifican las bacterias. Esta innovadora tecnología utiliza inteligencia artificial para convertir imágenes microscópicas de bacterias no teñidas en sus equivalentes teñidos de Gram, evitando así el proceso tradicional de tinción química. Los hallazgos han sido publicados en la revista Science Advances.
La tinción de Gram ha sido un pilar fundamental en microbiología durante más de un siglo, permitiendo diferenciar las especies bacterianas en dos grandes grupos (Gram-positivas y Gram-negativas) según las propiedades químicas y físicas de sus paredes celulares. La investigación, liderada por el profesor Aydogan Ozcan de UCLA, representa un avance significativo en los métodos de diferenciación bacteriana, abordando desafíos históricos de la tinción de Gram convencional, como la inconsistencia derivada de errores operativos y variaciones en la tinción química.
Tecnología y Beneficios de la Tinción Virtual
El sistema utiliza microscopía de campo oscuro para capturar imágenes de alta resolución de bacterias no teñidas, combinando esta técnica con tecnología de aprendizaje profundo para transformar virtualmente estas imágenes en sus equivalentes teñidos de Gram. Un modelo de red neuronal entrenado procesa un conjunto axial de imágenes de microscopía de campo oscuro, procesando rápidamente la información de dispersión óptica 3D de las células bacterianas para teñir digitalmente las bacterias sin etiqueta, igualando los resultados de la tinción de Gram tradicional.
El profesor Ozcan, autor principal del estudio, ha destacado: «La tinción de Gram tradicional, aunque fundamental para la microbiología, presenta limitaciones que pueden afectar la precisión diagnóstica. Nuestro enfoque de tinción virtual elimina estas variables, proporcionando resultados consistentes y rápidos sin la necesidad de reactivos químicos o de procesamiento manual de muestras por parte de expertos en microbiología.»
El equipo ha demostrado la efectividad de esta tecnología utilizando muestras bacterianas que contenían tanto bacterias Gram-positivas como Gram-negativas (Listeria innocua y Escherichia coli). Los resultados de la tinción virtual mostraron una alta precisión en comparación con el método tradicional, con los beneficios adicionales de reproducibilidad y estandarización.
Esta tecnología de tinción virtual ofrece numerosas ventajas para laboratorios clínicos, instalaciones de investigación e instituciones educativas. El sistema permite una diferenciación bacteriana rápida sin requerir pasos de procesamiento químico, agilizando significativamente el flujo de trabajo del análisis microbiológico. Al eliminar los procedimientos de tinción manual, se asegura que los resultados sean altamente consistentes y libres de variaciones relacionadas con el operador, abordando una limitación importante de los métodos tradicionales.
Además, esta tecnología reduce sustancialmente los costos operativos al eliminar la necesidad de tiempo de expertos humanos y de reactivos y materiales de tinción, convirtiéndola en una solución económicamente atractiva para instalaciones microbiológicas de todos los tamaños. Esta combinación de velocidad, fiabilidad y coste-efectividad hace que el sistema sea especialmente valioso en entornos donde la clasificación rápida y precisa de las bacterias es crucial para la toma de decisiones diagnósticas.
En un futuro prometedor, se vislumbra la posibilidad de análisis en tiempo real y tinción sin etiqueta de bacterias vivas. A diferencia de la tinción de Gram tradicional, que requiere fijar las bacterias, este enfoque de tinción virtual utiliza microscopía de campo oscuro que puede trabajar con organismos vivos sin necesidad de etiquetas. Los microbiológos podrían utilizar esta capacidad para estudiar bacterias sin etiqueta y revelar, por ejemplo, sus patrones de crecimiento y respuestas a antibióticos en tiempo real, sin interrumpir el estado natural de los microorganismos.
La investigación fue llevada a cabo por un equipo interdisciplinario de la UCLA, incluyendo departamentos de Ingeniería Eléctrica y de Computación, Bioingeniería, y el Instituto de Sistemas Nano de California (CNSI), en colaboración con investigadores del Departamento de Ingeniería Biomédica de Texas A&M y del Departamento de Patología y Medicina de Laboratorio de la Escuela de Medicina David Geffen de UCLA.