Un reciente estudio publicado en la revista hLife, liderado por el investigador Min Wang de la Universidad Oceánica de China, ha revelado importantes hallazgos sobre la diversidad de los virus que contienen transcriptasa inversa (RT). Este tipo de virus, que incluye a los retrovirus, desempeña un papel crucial en la replicación y en la mutación del genoma, lo que resulta en una amplia variedad de funciones biológicas. En el caso de los bacteriófagos, que son virus que infectan bacterias, la RT se utiliza no tanto para la replicación del genoma, sino para generar variaciones en los genes virales, lo que es esencial para la adaptación al huésped.
Los elementos generadores de diversidad (DGRs) son fundamentales para la adaptación de los bacteriófagos a sus hospedadores, especialmente en los ecosistemas intestinales de mamíferos. La capacidad de estos virus para promover mutaciones en genes relacionados con la adhesión al huésped, como las proteínas de cola y picos, les permite ajustarse mejor a las comunidades bacterianas en el intestino. A pesar de la diversidad funcional de las RT víricas, el conocimiento sobre ellas sigue siendo limitado.
Metodología y hallazgos del estudio
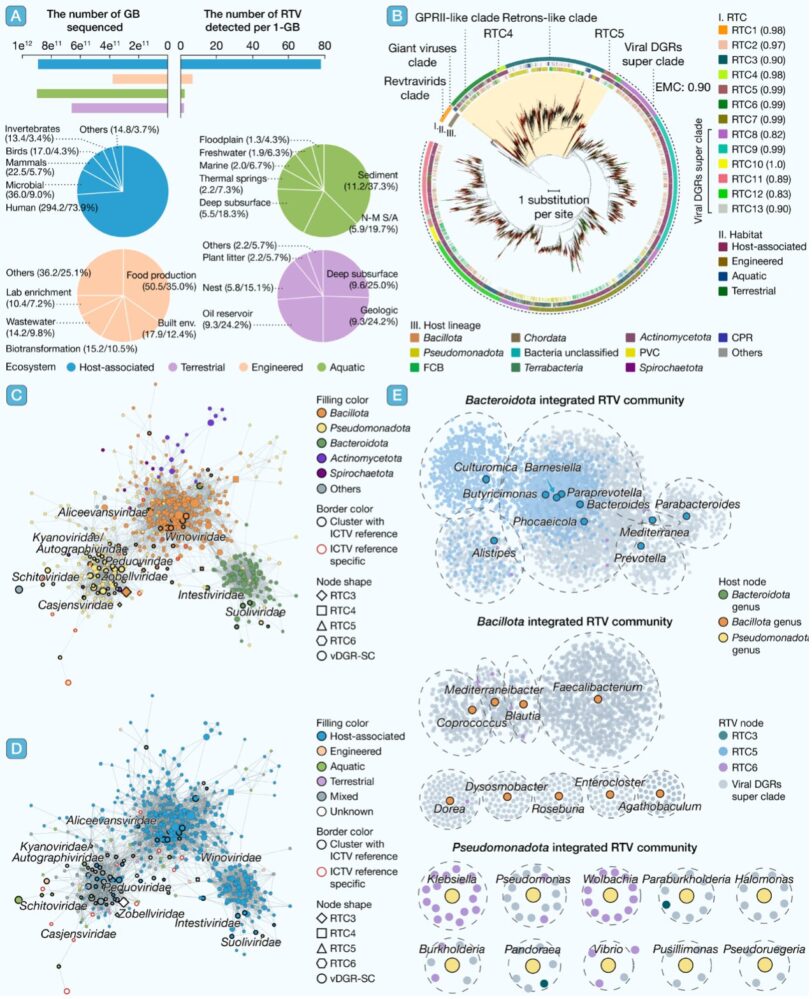
El equipo de investigación utilizó técnicas avanzadas de secuenciación de alto rendimiento y bioinformática para identificar 76,077 secuencias de RT víricas provenientes de 7,377 metagenomas globales. La distribución biogeográfica de estos genomas virales mostró su presencia generalizada en ambientes naturales, incluyendo suelos, aguas residuales, ecosistemas de agua dulce y marinos, así como su estrecha asociación con hospedadores animales. Un porcentaje significativo de estas secuencias de RT virales se derivó del intestino humano, lo que resalta el papel crítico de estos virus en el mantenimiento de la estabilidad de la microbiota intestinal humana.
Mediante un análisis filogenético riguroso, el estudio definió siete clados evolutivos de RT víricas, que incluyen clados de retrovirus, de virus gigantes, de DGRs de bacteriófagos, y otros. Estas agrupaciones monofiléticas están correlacionadas con sus funciones biológicas y rangos taxonómicos, lo que indica trayectorias evolutivas diversas y especializaciones funcionales de las RT víricas. La identificación de dos clados nuevos sugiere funciones aún inexploradas de las RT víricas, lo que podría tener implicaciones en el desarrollo de estrategias antivirales y aplicaciones biotecnológicas.
El estudio también identificó 78 grupos virales únicos compuestos exclusivamente por bacteriófagos que codifican RT. En la red de agrupamiento, estos bacteriófagos se agruparon en tres comunidades principales, cada una caracterizada por características de hospedador distintas. Las tres principales comunidades estaban asociadas con Bacillota, Pseudomonadota y Bacteroidota, respectivamente. Aunque la mayoría de los grupos estaban restringidos a entornos asociados al hospedador, 151 grupos se compartían entre diferentes hábitats, sugiriendo que ciertos bacteriófagos que codifican RT pueden migrar entre entornos a través de sus hospedadores infectados.
La integración de bacteriófagos que codifican RT en los genomas de los hospedadores a través de infecciones lisogénicas es un fenómeno común. Este estudio identificó numerosos profagos que codifican RT en los genomas completos o casi completos de 383 procariotas de 10 filos diferentes. La mayoría de estos profagos pertenecían al clado de DGRs de bacteriófagos y estaban ampliamente asociados con géneros de Bacillota, Bacteroidota y Pseudomonadota.
Entre estos, Bacteroidota fue identificado como la principal línea de hospedadores, comprendiendo 17 géneros y 4,461 profagos que codifican RT. Más de la mitad de estos profagos se compartieron entre diferentes géneros, lo que indica infecciones frecuentes entre géneros asociados a Bacteroidota. Dado el papel crucial de Bacteroidota en la microbiota intestinal humana, estos hallazgos sugieren que los bacteriófagos que codifican RT pueden contribuir a la adaptación de Bacteroidota al entorno intestinal.
Este estudio proporciona una exploración sistemática y un análisis exhaustivo de las RT víricas, revelando su diversidad evolutiva y funcional. Los hallazgos contribuyen al desarrollo de nuevas herramientas genéticas y objetivos terapéuticos, avanzando nuestra comprensión de las interacciones virus-microbios en el medio ambiente y destacando el papel de los virus en el mantenimiento de la estabilidad de la microbiota intestinal humana.