Investigadores de la Universidad de Zhejiang y de la Universidad de Ciencia y Tecnología de Hong Kong (Guangzhou) han desarrollado un modelo de inteligencia artificial de vanguardia llamado ProtET, que utiliza el aprendizaje multimodal para facilitar la edición controlada de proteínas mediante instrucciones basadas en texto. Este enfoque innovador, publicado en la revista Health Data Science, establece un puente entre el lenguaje biológico y la manipulación de secuencias de proteínas, mejorando el diseño funcional de proteínas en áreas como la actividad enzimática, la estabilidad y la unión de anticuerpos.
Las proteínas son fundamentales para las funciones biológicas, y su modificación precisa ofrece un potencial inmenso para terapias médicas, biología sintética y biotecnología. Mientras que los métodos tradicionales de edición de proteínas dependen de experimentos de laboratorio laboriosos y modelos de optimización de una sola tarea, ProtET introduce una arquitectura de codificador estructurada como transformador y un paradigma de entrenamiento jerárquico. Este modelo alinea las secuencias de proteínas con descripciones en lenguaje natural utilizando el aprendizaje contrastivo, lo que permite modificaciones intuitivas de proteínas guiadas por texto.
Avances en la Edición de Proteínas
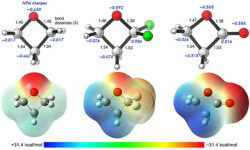
El equipo de investigación, liderado por Mingze Yin de la Universidad de Zhejiang y Jintai Chen de la Universidad de Ciencia y Tecnología de Hong Kong, entrenó a ProtET con un conjunto de datos de más de 67 millones de pares de proteínas y biotextos, extraídos de las bases de datos Swiss-Prot y TrEMBL. El modelo demostró un rendimiento excepcional en varios benchmarks, mejorando la estabilidad de las proteínas en hasta un 16,9% y optimizando las actividades catalíticas y la unión específica de anticuerpos.
«ProtET introduce un enfoque flexible y controlable para la edición de proteínas, permitiendo a los investigadores ajustar funciones biológicas con una precisión sin precedentes», afirmó Mingze Yin, autor principal del estudio.
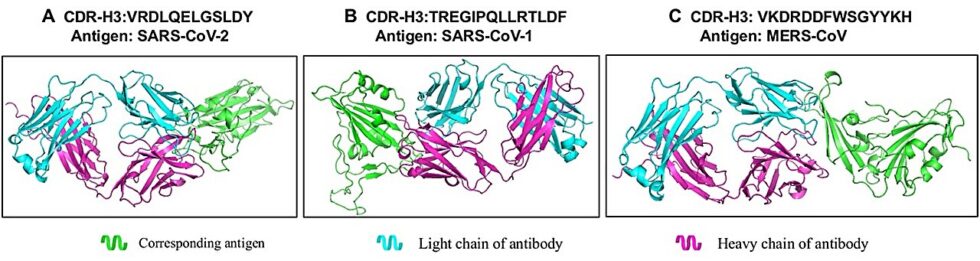
El modelo logró optimizar secuencias de proteínas en diferentes escenarios experimentales, incluyendo la actividad catalítica enzimática, la estabilidad de las proteínas y la interacción de unión entre anticuerpos y antígenos. En tareas de cero disparo, ProtET diseñó anticuerpos contra el SARS-CoV que formaron estructuras 3D estables y funcionales, demostrando su aplicabilidad en la investigación biomédica.
De cara al futuro, el equipo prevé que ProtET se convierta en una herramienta estándar en la ingeniería de proteínas, allanando el camino para avances en biología sintética, terapias genéticas y manufactura de biofármacos. Este estudio representa un paso transformador en el diseño de proteínas impulsado por inteligencia artificial, mostrando cómo la integración multimodal puede desbloquear nuevos horizontes en el descubrimiento científico y la innovación.
Más información:
Mingze Yin et al, Multi-Modal CLIP-Informed Protein Editing, Health Data Science (2024). DOI: 10.34133/hds.0211
Proporcionado por
Health Data Science