Más de un centenar de expertos en simulación molecular han publicado un artículo en la revista Nature Methods que aboga por un cambio de paradigma en la gestión de datos de dinámica molecular. Este trabajo, liderado por Modesto Orozco, profesor de la Universidad de Barcelona, y el experto Adam Hospital, ambos miembros del Instituto de Investigación en Biomedicina (IRB Barcelona), propone la creación de una infraestructura común para almacenar y reutilizar datos en el contexto de la revolución que representa la inteligencia artificial.
El artículo defiende la implementación de principios FAIR (en inglés, findable, accessible, interoperable, reusable) para mejorar la reproducibilidad de los cálculos y facilitar su posterior uso como fuente de información sobre la flexibilidad de biomacromoléculas. Las simulaciones computacionales se han convertido en una herramienta clave para estudiar el comportamiento de biomoléculas a lo largo del tiempo. Gracias a los superordenadores, la dinámica molecular permite observar estos procesos con gran precisión y proporciona nuevos conocimientos de interés tanto en la investigación básica como en el diseño de biomoléculas, desde enzimas hasta fármacos.
A diferencia de la biología estructural o la genómica, donde el almacenamiento y el intercambio de datos bajo estándares comunes son prácticas habituales, en el ámbito de la simulación molecular estos datos permanecen fragmentados. A menudo terminan olvidados en ordenadores personales, lo que obstaculiza la reproducibilidad de los cálculos y impide su uso posterior. Esta situación plantea un gran problema en la integración de datos en flujos de trabajo de biología estructural y biofísica, además de ralentizar el desarrollo de métodos de inteligencia artificial, los cuales dependen en gran medida del acceso a grandes volúmenes de datos dinámicos.
Promoviendo la reutilización de datos
El objetivo del nuevo artículo, respaldado por más de un centenar de investigadores internacionales de renombre, incluidos varios premios Nobel de química, es diseñar un ecosistema abierto y sostenible que multiplique el impacto de estos datos y evite duplicaciones innecesarias. Los autores llaman a un cambio de modelo que aplique los principios FAIR a los resultados de simulación. Como señala el Dr. Orozco, «la comunidad ha asumido durante años que repetir una simulación era más fácil y barato que archivarla. Pero eso ya no es cierto».
La reutilización de datos puede proporcionar un enorme valor, permitiendo identificar nuevos objetivos, entrenar algoritmos de inteligencia artificial o diseñar nuevos experimentos. Orozco y Hospital lideran el proyecto europeo MDDB, que tiene como objetivo establecer una base de datos centralizada y accesible para simulaciones. Se inspiran en el éxito de otros campos que han adoptado la ciencia abierta, como el Protein Data Bank, que ha recopilado estructuras tridimensionales de biomacromoléculas desde los años 70 y ha sido fundamental para el desarrollo de fármacos, vacunas y nuevas terapias.
Los datos almacenados en esta base fueron clave para el entrenamiento de AlphaFold2, que recibió el Premio Nobel de Química en 2024. Los autores del artículo sostienen que complementar estos datos estructurales con información dinámica abrirá un nuevo campo de potencial de desarrollo difícil de calcular.
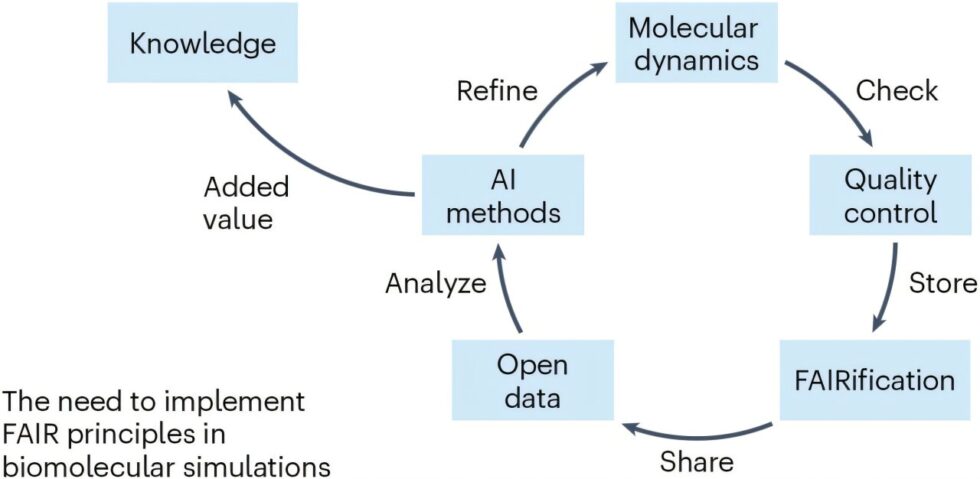
Los autores consideran que ha llegado el momento de que la comunidad de simulación molecular adopte prácticas similares a las de las comunidades estructurales y «ómicas», no solo preservando datos, sino también estandarizando formatos de archivo, metadatos y criterios de calidad. El texto describe cómo una infraestructura federada, con nodos distribuidos y herramientas de acceso compartido, podría hacer viable este archivo a escala planetaria.
La propuesta va más allá del simple almacenamiento de datos, abogando por un modelo integrado que contemple desde la documentación precisa de simulaciones (incluyendo condiciones, software, parámetros, etc.) hasta su análisis automatizado, validación y reutilización mediante técnicas de aprendizaje automático. Para el Dr. Orozco, «el valor de estos datos no termina con la publicación de un artículo o su presentación en una conferencia. A menudo, eso es solo el comienzo». Por ello, enfatiza la necesidad de tratar los datos como un recurso compartido para la ciencia.
Este artículo ha sido elaborado en el marco del Proyecto Europeo MDDB (Molecular Dynamics Data Bank), coordinado por IRB Barcelona, que busca construir una base de datos abierta y estandarizada para almacenar simulaciones moleculares dinámicas. El consorcio reúne a centros de investigación líderes en bioinformática, simulación y análisis de datos para avanzar hacia una ciencia más abierta, reproducible y colaborativa.