Investigadores del Instituto Conjunto de Genómica (JGI) del Departamento de Energía de EE. UU., ubicado en el Laboratorio de Berkeley, han publicado un estudio en Science Advances que evalúa el estado actual de la biodiversidad microbiana genómica. Utilizando datos de secuencias genómicas disponibles públicamente generados a lo largo de los últimos treinta años, el estudio analiza qué fracción de la diversidad microbiana conocemos y propone un camino a seguir para curar y cultivar lo que aún permanece desconocido.
Según Dongying Wu, coautor principal del estudio, «realizamos un análisis profundo de más de 1.8 millones de genomas bacterianos y arqueales para ver cuánta diversidad hemos capturado realmente». A pesar de los esfuerzos realizados en la secuenciación, el equipo concluyó que «solo hemos arañado la superficie». Esta investigación sirve como un llamado a revitalizar la microbiología experimental y la validación práctica.
Los microbios desempeñan un papel crucial en la regulación de los ciclos de nutrientes globales. Comprender las interacciones entre microbios, huéspedes y sus entornos podría aplicarse a múltiples áreas de investigación, incluyendo la agricultura, los biocombustibles, los bioproductos y la medicina. El equipo, que también incluye a Rekha Seshadri, Nikos Kyrpides y Natalia Ivanova, realizó un censo de las secuencias bacterianas y arqueales disponibles públicamente. Kyrpides dirige el grupo de Ciencia de Datos del Microbioma del JGI, mientras que Ivanova lidera el equipo de Anotación Funcional.
Ampliación significativa de la diversidad estimada de Bacterias y Arqueas
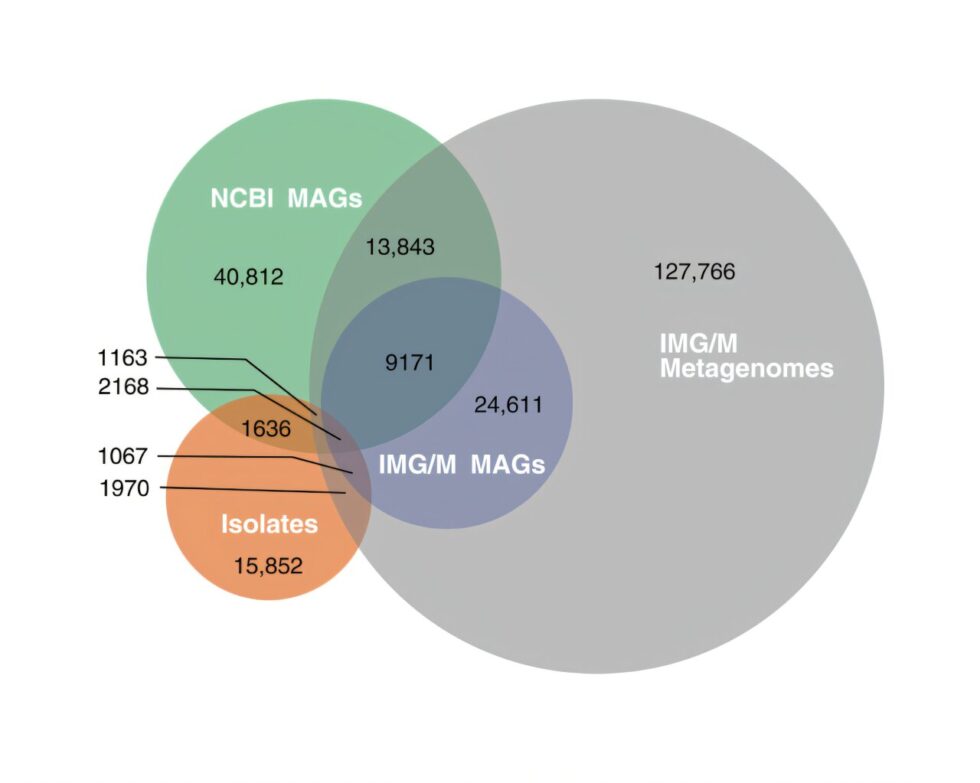
Utilizando la diversidad filogenética como medida representativa de biodiversidad, los investigadores emplearon cinco genes marcadores de proteínas universalmente conservados sobre casi dos millones de genomas, incluyendo genomas aislados y genomas ensamblados a partir de metagenomas (MAGs). Todos los datos están disponibles públicamente en la colección GenBank del Centro Nacional de Biotecnología de EE. UU. y en la base de datos Integrada de Genomas y Microbiomas del JGI (IMG/M).
En su análisis, el equipo descubrió que los genomas aislados bacterianos representan solo el 9.73% de la diversidad total estimada de los conjuntos de datos disponibles. Los esfuerzos para recuperar MAGs mediante la extracción de datos directamente de muestras ambientales han ampliado significativamente la diversidad conocida de los genomas microbianos, representando casi el 49% de la diversidad bacteriana total estimada. Se estima que aproximadamente el 42% de la diversidad bacteriana no tiene representación genómica en las bases de datos públicas.
En un análisis similar para las Arqueas, el equipo encontró que los genomas aislados representan solo el 6.55%, mientras que los MAGs contabilizan alrededor del 57% de la diversidad estimada de los conjuntos de datos disponibles. Esto deja un 36% de la diversidad arqueal sin representación genómica.
La investigación subraya la necesidad urgente de cultivar nuevas especies microbianas. Ivanova afirmó que el JGI sigue trabajando para aumentar la representación genética de la diversidad bacteriana y arqueal, iluminando el espacio microbiano desconocido. Aunque los MAGs han ampliado significativamente la diversidad conocida de genomas microbianos, el equipo advierte que esta información sigue siendo derivada computacionalmente. Se requieren estudios experimentales sobre aislamientos cultivados para convertir las implicaciones potenciales en ciencia aplicada, contribuyendo así hacia una bioeconomía sostenible.
«Mientras que los MAGs derivados computacionalmente han sido una herramienta revolucionaria para la microbiología, es un llamado a encontrar un equilibrio», añadió Seshadri. Destacó que los conjuntos de datos metagenómicos utilizados en este estudio podrían ayudar a los investigadores a mejorar sus posibilidades de recuperar especies aisladas específicas para cultivo. «Hemos trazado un mapa del tesoro», afirmó. «Básicamente, podemos señalar específicamente a muestras ambientales donde las personas pueden reinvertir tiempo y esfuerzo en la recuperación».
Más información:
Dongying Wu et al, A metagenomic perspective on the microbial prokaryotic genome census, Science Advances (2025). DOI: 10.1126/sciadv.adq2166